Elena Canorea
Communications Lead
Durante los últimos años, se ha realizado un esfuerzo de investigación con el objetivo de poder automatizar el análisis de imágenes médicas empleadas en el ciclo diagnóstico-terapéutico.
En la medicina moderna, realizar diagnósticos utilizando imágenes es invaluable. El proceso de imágenes de Tomografía Computarizada (CT – Computer tomography) así como otras modalidades, proveen un medio no invasivo y efectivo para delinear la anatomía del sujeto.
La segmentación de imágenes se ha convertido en un proceso clave para el delineamiento de ciertas estructuras anatómicas y otras regiones, con el objetivo de asistir y ayudar a los médicos en cirugías, biopsias y demás pruebas clínicas.
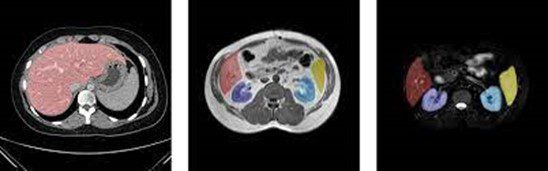
Ejemplo de segmentación de órganos
A lo largo del tiempo, las técnicas de Inteligencia Artificial, y en concreto, el uso de “Deep Learning”, han permitido que el campo de visión por computación avance rápidamente en los últimos años. En este artículo, mostramos como podemos utilizar técnicas de Segmentación semántica para la detección de órganos en imágenes médicas.
Actualmente, las redes neuronales convolucionales se han utilizado para resolver distintas tareas dentro del dominio de Visión Artificial. La resolución de cada una de estas tareas nos aporta un grado de comprensión distinto de las imágenes.
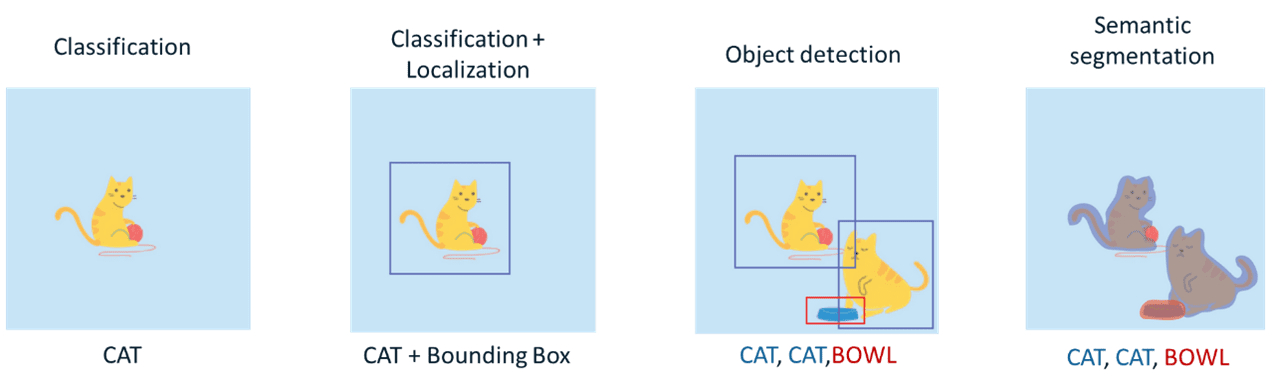
Tipos de tareas Computer Vision
Como ya se ha comentado anteriormente, en este caso nos centraremos en una de las tareas más complejas dentro del campo de visión por computación: La Segmentación semántica. El objetivo de esta tarea (dentro del ámbito de las imágenes) es etiquetar cada píxel de una imagen con la clase correspondiente de lo que está representando. Debido a que estamos realizando la predicción para cada píxel de la imagen, esta tarea es comúnmente conocida como predicción densa.
El rendimiento que obtenga nuestro modelo dependerá en gran parte de la cantidad y la calidad del conjunto de datos. Por este motivo, la selección de este es uno de los procesos más importantes dentro del flujo de trabajo de proyectos de Machine Learning.
Tal y como se ha mencionado al principio del artículo, el objetivo de nuestro modelo es detectar órganos (a nivel de segmentación semántica) a partir de imágenes médicas del Abdomen.
Debido a la sensibilidad de esta información médica, se ha utilizado un conjunto de datos (CHAOS (Combined Healthy Abdominal Organ Segmentation) Dataset) anonimizado y el cuál se usa específicamente para temas de investigación dentro de esta área. Este conjunto de datos está compuesto por diversas fuentes. En este caso, nos hemos centrado en utilizar un subconjunto de datos para la detección de los siguientes órganos: Bazo, riñones (izquierdo y derecho) e hígado.
A lo largo de este apartado, se comentarán los aspectos más importantes que se han llevado a cabo durante nuestro proceso de entrenamiento para la segmentación de órganos.
Como nuestro conjunto de datos está formados por imágenes DICOM, antes de lanzar el proceso de entrenamiento, es necesario procesar todos nuestros datos y convertir dichas imágenes en array de píxeles. Una vez que se ha hecho la extracción de dicho array, se realizarán varias transformaciones: afinidad y reducción del tamaño de la imagen. Por último, las imágenes se normalizan con el objetivo de obtener un mejor rendimiento durante la fase de entrenamiento.
U-net es una arquitectura de red neuronal convolucional que fue desarrollada para la segmentación de imágenes biomédicas, por lo tanto, nos basaremos en su arquitectura base para las definiciones de nuestros modelos. En esta ocasión, hemos utilizado algunas variantes de la topología original para realizar nuestro proceso de entrenamiento: U-Net con (Resnet18) y U-Net++.
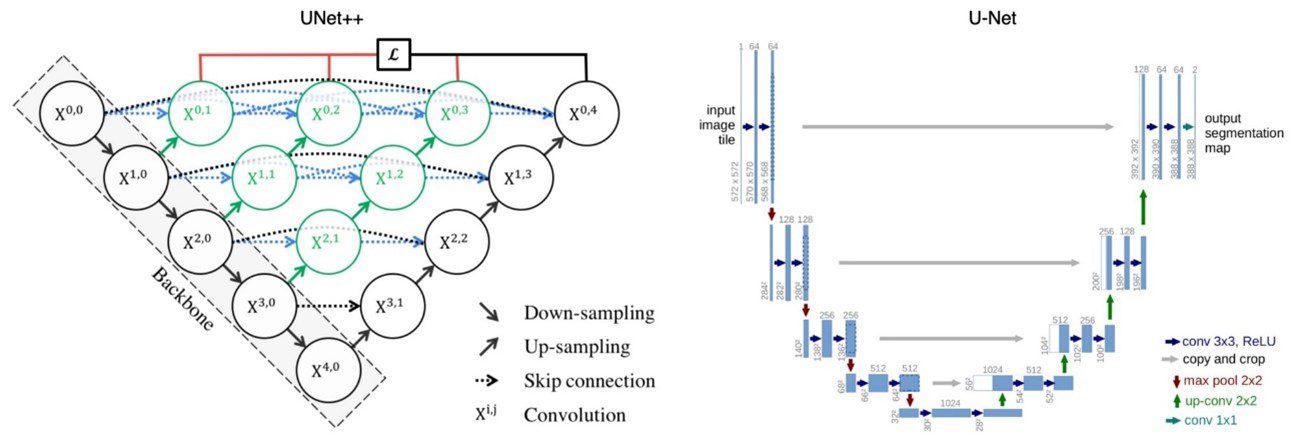
Topología de los modelos utilizados
La función de pérdida en el ámbito de las redes neuronales es una función que evalúa la desviación entre las predicciones realizadas por la red y los valores reales de las observaciones utilizadas durante el proceso de entrenamiento. Cuanto menor es el resultado de esta función, más eficiente es la red neuronal. El objetivo del entrenamiento de una red neuronal es intentar minimizar lo máximo posible esta función para reducir al mínimo la desviación entre el valor predicho y el real.
Una de las funciones de pérdidas más utilizadas en el caso de segmentación de imágenes es el coeficiente “dice”. Este coeficiente nos sirve para conocer el grado de solapamiento que hay entre la máscara predicha y la máscara etiquetada.
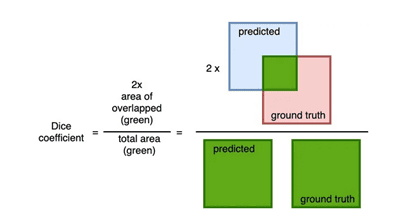
Cálculo del coeficiente «dice»
El coeficiente “dice” tiene un rango de valores entre [0,1] donde 1 indica un solapamiento total, por lo tanto, si queremos minimizar al máximo posible esta función, el valor de (1- Dice cofficient) puede ser utilizado como función de pérdida.
En nuestro caso, la función de pérdida final que se ha utilizado para el entrenamiento es una combinación entre la función de pérdida Weighted Binary Cross Entropy y la función de perdida dice
En esta sección, comentaremos los resultados que se han obtenido durante el proceso de entrenamiento.
Los hiperparámetros que se utilizaron en el entrenamiento fueron los siguientes:
Tal y como podemos observar en la siguiente imagen, el modelo de Unet-Resnet converge más rápido que el modelo de Unet++ (esto se debe a la utilización de un modelo con pesos pre-entrenados en las capas de “encoder” y a un menor número de parámetros).
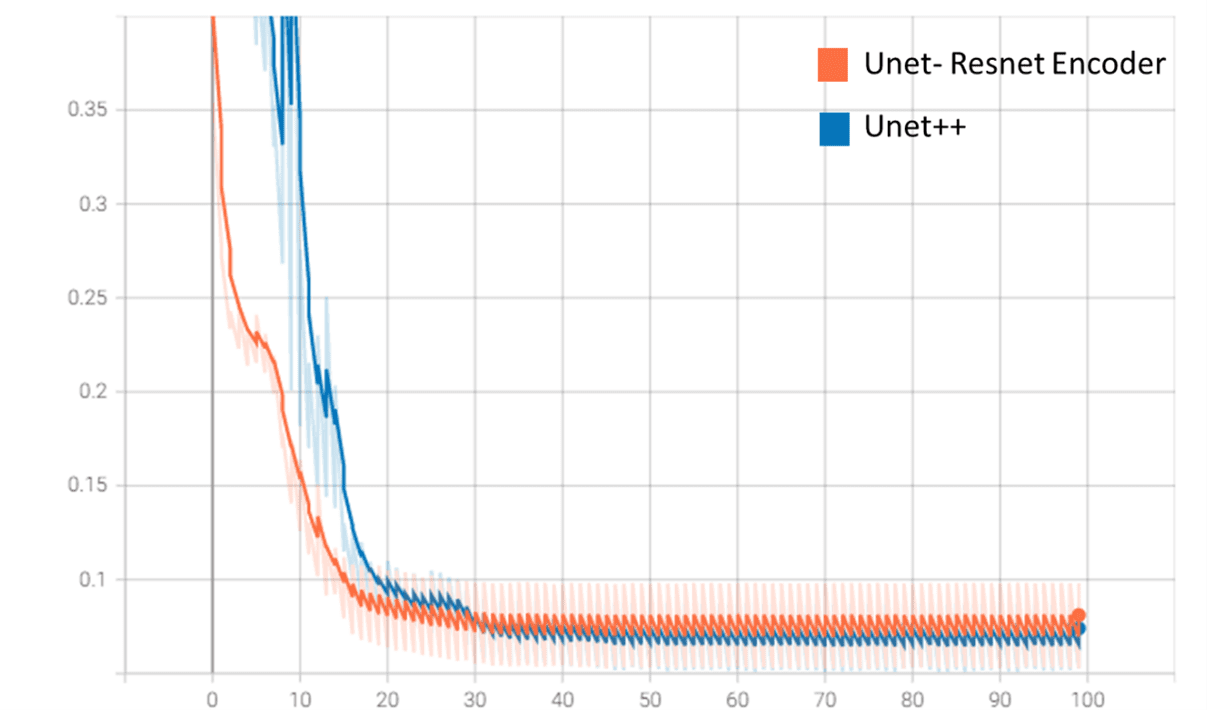
Valores función de pérdida (validación) en Tensorboard
Por otro lado, aunque el modelo de Unet++ obtiene un mejor rendimiento en validación (función de pérdida “dice”), Unet-Resnet entrena mucho más rápido y tiene un tamaño menor (70 MB frente a los 500 MB de Unet++)
A continuación, se muestran varios resultados del modelo utilizando el conjunto de datos de validación. Cada ejemplo está formado por: la imagen de entrada, la imagen etiquetada por el médico/especialista y la predicción realizada por el modelo.
Código de colores: Naranja- Bazo, Amarillo – Riñón derecho, Marrón – Riñón Izquierdo, Azul- Hígado
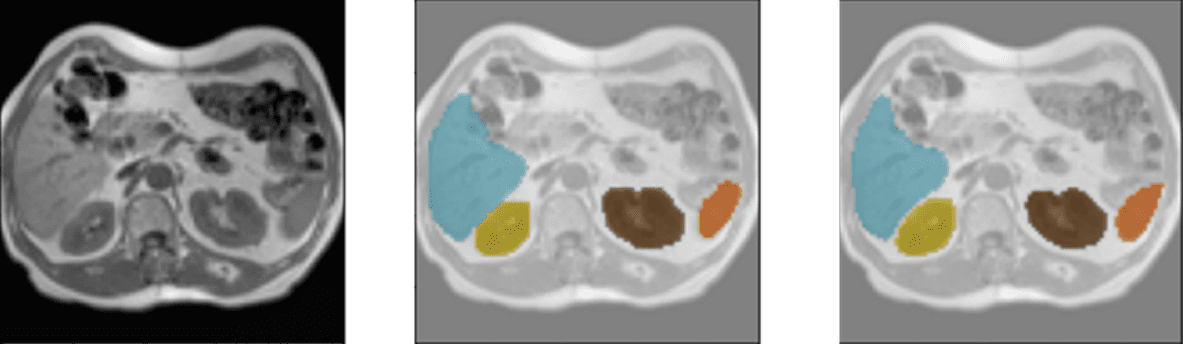
Detección de hígado, bazo y riñones
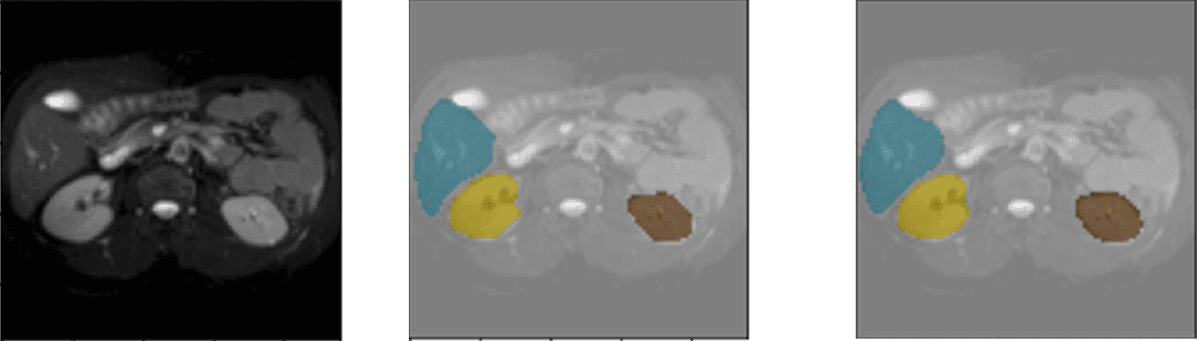
Detección de hígado y riñones
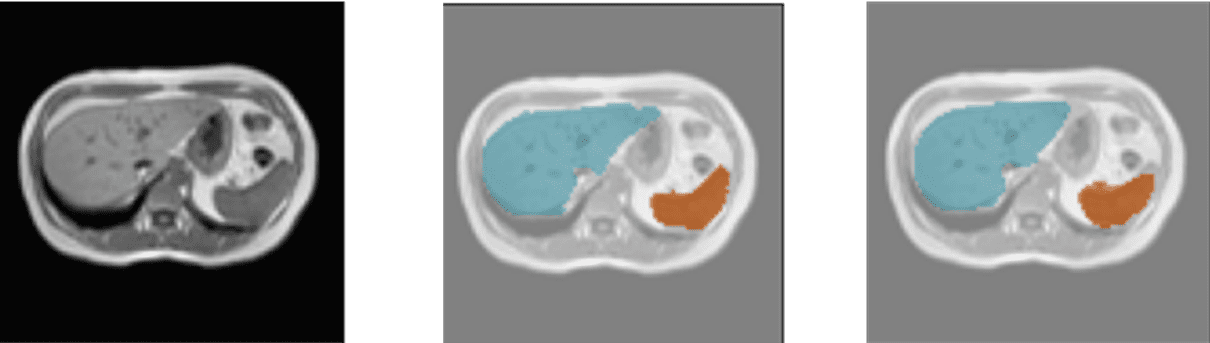
Detección de hígado y bazo
El uso de técnicas de Deep Learning en imágenes médicas puede ayudar a los especialistas en la toma de decisiones a la hora de realizar un diagnóstico.
Una de las primeras tareas que pueden facilitar el uso de este tipo de técnicas, es la integración del modelo en cualquiera de los dispositivos hardware que utilicen los especialistas para visualizar o representar imágenes médicas (dispositivos móviles, HoloLens y ordenadores). Para llevar a cabo este proceso, será necesario evaluar previamente el rendimiento del modelo y aplicar distintas técnicas de optimización tales como: “Pruning o cuantización”. La aplicación de estos métodos nos asegurará que nuestro modelo se pueda adaptar mejor a las posibles limitaciones hardware del dispositivo.
Tal y como hemos visto en este artículo, nuestro modelo es capaz de detectar órganos a partir de imágenes médicas en 2D. Como posible trabajo futuro, se podría integrar este modelo dentro de dispositivos que permitan interactuar con una representación en 3D del abdomen.
El uso de nuestro modelo dentro de estas soluciones ayudará a mejorar gran variedad de pruebas clínicas. El sistema puede mostrar en todo momento donde están ubicados los órganos y así, poder planificar la punción óptima que produzca el menor número de daños a los órganos adyacentes.
Elena Canorea
Communications Lead
